基于U-Net的直肠癌CT图像分割系统的设计与实现
作者: 许晶淼 胡天寒 刘影 吴敏 林莉
关键词:直肠癌;语义分割;深度学习;U-Net 网络
中图分类号:TP183 文献标识码:A
文章编号:1009-3044(2023)32-0008-03
0 引言
近年来在我国乃至全世界,直肠癌的发病率与死亡率都高于其他恶性肿瘤。而目前,大多数医院依靠放射科医生人工标注直肠肿瘤区域,这种方法依托于医生的主观性,因此利用深度学习在直肠癌CT图像上的分割,可以给医生提供临床诊断上的参考意见,提高诊断效率。
机器学习技术在国外多领域发展,该技术也被应用于研究直肠癌放射学领域,使得在医学图像研究领域成果显著增加。Ran G等[1]基于决策树模型,以年龄、性别以及全血计数作为特征,来检测患者早期结直肠癌;Bychkov D等[2]利用机器学习技术,将深度学习模型应用于预测患者结直肠癌的预后,结果显示该模型可提取的预后信息比具有一定工作经验的医生要更多;Li Y等[3]基于术前CT 图像的数据,开发了一个机器学习模型,用于预测在结直肠癌中肝转移的情况,该模型可以为高危直肠癌患者出现肝转移的情况下提供个性化的监测;Trebeschi等[4] 将卷积神经网络训练在磁共振弥散加权图像上,该研究可以自动定位并分割直肠癌,但是数据量太小,无法支撑网络的训练。
在国外研究成果的引领启发下,我国将深度学习技术应用于直肠癌分割的研究也越来越多。南京医科大学戴薇等[5]研究通过采用基于U-Net的自动分割模型对直肠癌GTV和OARs进行勾画,实验结果显示对OARs勾画准确性较高,但是对GTV勾画的准确性仍需提高。华南师范大学肖波等[6] 使用240张直肠癌CT 图像进行训练与测试,利用FCN-8s、U-Net 以及SegNet模型对直肠癌CT图像进行分割,以此来定位病灶位置,实验结果表明,U-Net模型和InceptionV3 模型分割结果更好,分类准确率高。西南科技大学汪豪等[7]提出能有效避免梯度消失问题的一种基于残差块端到端改进的U型神经网络模型,该模型结合图像裁剪预处理,可提高直肠癌的分割精度。
本文提出了一种改良卷积层的U-Net模型,对直肠癌CT图像进行分割以及系统实现。基于卷积神经网络对直肠和肿瘤区域进行分割,使用在临床试验上应用最多的U-Net模型,对其卷积块间增加归一化层构成新的Conv-concat模块。改进后的U-Net模型在直肠癌CT图像上对肿瘤分割的精确率显著提高,在直肠癌临床诊断中可提供有价值的参考信息,以制定治疗方案。
1 数据集和模型设计
1.1 数据集的选取
本文使用第7届“泰迪杯”数据挖掘挑战赛中的数据集。该数据集包含2部分,一部分是未标注的直肠癌CT图像的train数据,另一部分是已标注好对应的直肠癌掩膜的label数据。train数据中包含108位患者的CT图像文件夹,每位患者有20~30张CT图像,原图像为DCM格式,共计3 166张CT图像,其中精选3 000幅CT图像用于实验分割。
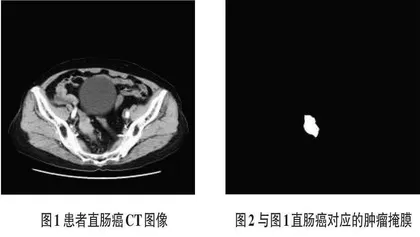
任取一名患者直肠癌CT图像如图1所示,以及该患者直肠癌对应掩膜如图2所示。
1.2 图像预处理
首先,读取数据集,再结合本文数据集的特点,选择图片剪裁与图像归一化这2种预处理方法。
1)读取数据
由于train数据集中各个文件夹的CT图像数量不一样,所以需要使用Python中glob()函数读取文件夹和文件名,之后再使用Image.open()函数读取图片。
2)图片裁剪
直肠肿瘤只发生在直肠,实际需要处理的区域并不需要整张图片,适当裁剪能够提高网络训练的速度。图3为使用图片裁剪技术后的效果图。
3)图像归一化
使用标准归一化是数据处理中一个重要步骤,此方法可有效地减少图像数据中一些异常值对实验的影响。神经网络利用梯度下降的方法来反向传播更新误差,尤其在数据差值很大的情况下,标准归一化非常重要。
1.3 训练模型
U-Net整体的网络结构表现为对称的U型,其设计主要针对于医学图像的分割,并且U-Net网络因其不错的训练效果、较高的训练效率和可以适应很小的训练集等特性引起了许多研究者的关注。
1.3.1 U-Net 模型介绍
U-Net网络是一种可以实现端到端映射的网络,可实现图像分割、恢复、增强和超分辨等目标。其具体网络结构如图4所示。本文对网络输入256×256大小的图像进行初始特征提取,网络可分为左右两部分,左边部分是收缩路径,使用降采样和卷积模块提取不同尺度的特征;右边部分是扩展路径,使用上采样和卷积模块恢复尺度并融合先前特征,恢复图像并输出。
卷积模块由两层连续的卷积层组成,其中卷积核的大小设置为3×3,步长为1,Padding为same,以此来实现更大尺寸、更高深度的特征提取;降采样实现图像尺度的缩小;上采样(或反卷积层)实现图像尺度的变大;图中灰色的箭头线表示跳连,在同一尺度下,将收缩路径上的特征合并到扩展路径上的特征。
1.3.2 改进U-Net 模型卷积层
本文在传统U-Net卷积块(如图5所示)间增加归一化层,构成了新的Conv-concat模块(如图6所示)。相比于未改进的U-Net模型的卷积块,Conv-concat模块做了两点改进。
首先,让卷积产生的特征图和最开始输入的Conv-concat模块的特征图进行整合,将整合的结果作为下一次卷积的输入。其次,为了降低网络模型对初始化参数的依赖,在Conv-concat模块中,在每一个卷积层后都增加一个BN层(即Batch Normalization)对输入进行标准化,使得将每一批次中的数据正态化。
2 实验设计与结果分析
2.1 实验设计
为了验证U-Net模型对直肠癌CT图像分割的效果,在共计3 000张图片的数据集上进行实验,因为肿瘤分布是连续的,为了避免局部特征过于集中,对数据集进行乱序,之后再按9:1分配,其中包括训练集2700张和验证集300张。
实验在型号为NVIDIA GTX 2060的GPU上进行训练。U-Net网络模型输入的图片尺寸为256×256×1,训练的轮次分别设置为100次,200次和400次,使用Adam优化器和均方误差损失函数。
2.2 评估标准
本实验使用Dice系数、准确率Acc作为评估标准来检测模型的性能。
1)Dice系数
对图像分割的评价首先采用Dice系数,其中A是医生勾画的直肠肿瘤区域,B是算法分割后获得的直肠肿瘤区域。将Dice系数的取值范围设置在[0,1],若取值越接近1,则表明经网络训练得到的直肠肿瘤分割结果与医生人工标注的结果越接近,即分割结果较为精确。Dice系数计算公式如式(2)所示:
2)准确率Acc
本文将正确分割直肠癌CT图像记为正类,未分割出直肠癌位置或错误分割记为负类。将正类中检测成正类数记为TP,正类中检测成负类数记为FN;将负类中检测成正类数记为FP,负类中检测成负类记为TN。混淆矩阵如表1所示:
2.3 实验结果与分析
以下是初步实验得到的结果图,results保存了每一轮训练集的loss和metric以及验证集的val_loss和val_metric,将其绘制出来,得到的曲线如图7所示。
从图7中结果可以看出,验证集的损失值总是比训练集大,即肿瘤分割效果总比训练集差,说明评价网络要用未训练的数据进行测试,网络需要通过不断地拟合训练集,以此达到最佳的训练效果。本次试验中,验证集的Dice系数达到了0.82,这是一个比较高的值,说明网络训练效果尚可。
针对本文的肿瘤分割问题,评价指标主要有2个,一是分割区域精确度,也就是Dice系数;二是肿瘤分割的准确率,即Acc。
在300张测试集中,分割的直肠肿瘤平均Dice系数达到了0.821 2,表明分割的肿瘤区域与医生给予的掩膜较为符合。计算得出测试集的准确率Acc达到了99.44%,这表明网络对于有肿瘤图片和没有肿瘤图片的区分是非常准确的。
2.4 基于U-Net 的直肠癌CT 图像分割系统的实现
将训练的最优模型提取出来并设计直肠癌分割系统。医生上传直肠癌CT图像,系统输入端获取直肠癌CT图像,通过模型处理并输出生成相应的直肠癌病灶的位置和大小。具体系统界面如图8所示,先通过“上传图片”,选取要分割的图像,点击“检测结果”,即在右边得到直肠癌病灶位置和大小。
3 总结
本文使用深度学习中U-Net网络,并基于该网络模型改进其卷积块部分,对直肠癌肿瘤区域进行分割并实现分割系统。实验结果测试集上的准确率Acc 达到了99.44%,可以准确区分有无肿瘤的CT图像,并且能给予一些辅助性诊断参考意见。但是分割的肿瘤平均Dice系数为0.8212,表明分割的肿瘤区域与医生给予的掩膜符合度仍需提高,预测的肿瘤区域轮廓不够细致。