基于RBBEGP的中文电子病历命名实体识别研究
作者: 郭振华 宋波
关键词:中文电子病历;命名实体识别;RoBERTa-wwm-ext;BiGRU;EGP
0 引言
随着我国医疗信息化的快速发展,中文电子病历数据也在不断积累。电子病历作为病人信息的医疗服务健康记录,在医疗领域中发挥着重要的作用。由于其以非结构化文本形式存储,导致数据难以被理解与表示,限制了其发展与应用。命名实体识别(NamedEntity Recognition, NER) 是一种信息抽取技术,能够从非结构化文本中自动识别出特定实体并进行归类[1]。NER的发展经历了从基于规则的传统方法到机器学习方法,再到深度学习方法的演变[2],目前已被广泛应用于各类自然语言处理(Natural Language Pro⁃cessing, NLP) 任务中[3]。通过使用NER技术从电子病历文本中准确地提取疾病、诊断、药物等医疗类别,可以更高效地分析和处理电子病历中的信息。
当前,国内外专家学者不断改进深度学习模型以提高命名实体识别的效果。Devlin等人提出的预训练模型BERT(Bidirectional Encoder Representation fromTransformers) 在多种NLP任务中通过微调取得了较好的效果[4]。LEE等人在BERT的基础上提出了针对于生物医学领域的BioBERT(Biomedical BERT) ,该模型在生物医学领域中的命名实体识别、医学关系提取及问答等任务上F1 值分别提升了0.62%、2.80%、12.24%[5]。上述模型均以字为单位进行切分,然而在中文语境下,往往通过一个词来表示完整的含义。为此,哈工大讯飞联合实验室提出了RoBERTa-wwmext[6],该模型能够注重整个词的含义, 更适合于中文语料的训练任务。在命名实体识别模型中,常采用CRF(Conditional Random Field) [7]对序列标注进行预测,但中文电子病历文本中涉及到解剖结构、疾病分类、治疗方法等复杂的医疗类嵌套命名实体,这些实体之间有着较深的层级关系,以致CRF在医疗领域的命名实体识别任务中表现欠佳。2022年,Su等人提出了全局指针网络模型(Global Pointer, GP) 及其改进版EGP,结果表明该模型相较于CRF能够更有效地识别嵌套实体,且对非嵌套实体的识别效果也与CRF 相当[8]。
综上所述,传统的命名实体识别模型在中文电子病历上的识别效果局限于两个方面:一是模型未能考虑中英文字词间的表述差异;二是在嵌套命名实体较多的情况下难以准确识别。针对以上问题,本文提出了一种基于RoBERTa-wwm-ext-BiGRU-EGP(RBBEGP) 的命名实体识别模型。该模型能够准确地解析命名实体,从而进行医学信息提取和文本分析[9],帮助医疗领域的相关人员理解和处理医疗文本数据。其中,预训练语言模型RoBERTa-wwm-ext有效解决了传统模型对中文文本识别准确率较低的问题;BiGRU能够获取病历上下文信息,并减少参数量以加快训练速度[10];最后,引入EGP以提高模型识别嵌套命名实体的能力。
1 相关工作
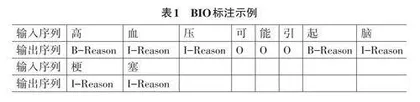
BIO标注法能够传递有限且精确的信息,有利于模型理解文本内容[11]。在中文电子病历文本中,一个序列对应的是文本中的一句话,而元素对应其中的每一个字。B(Begin) - α 表示此元素属于α 类实体且为开头位置,I (Inside) - α 表示此元素属于α 类实体且为实体的中间或结尾位置,O(Outside)代表此元素不属于任何已定义的实体类型。当输入文本序列“高血压可能引起脑梗塞”时,经BIO序列标注后的输出如表1所示。
命名实体识别模型主要由词嵌入层、编码层和解码层3个部分组成。
词嵌入层使用预训练语言模型(Pretrained Lan⁃guage Models, PLMs) ,有利于学习语言的潜在结构和规律,并从中抽象出通用的语言表示形式。PLMs 指的是在训练过程中先使用大量无标签的数据对模型进行初步训练,使其具备一定程度的通用语言理解能力,然后再针对特定任务对模型进行微调。这种训练方式使得模型可以有效地对词的多义性进行建模,表达出丰富的句法和语法信息,从而在医疗领域等各类下游任务中获得更好的性能表现。
编码层将输入的文本序列转化为模型可以处理的内部表示形式。编码器的主要任务是学习输入文本的语义特征,将文本中的词语或字符映射到一个高维的向量空间中,以便于模型进行进一步的处理和识别。双向长短期记忆网络[12] (Bi-directional LongShort-Term Memory,BiLSTM) 作为一种常用的编码器,能够对输入的语义信息选择性地遗忘或传递,从而获取长序列文本依赖的特征信息。然而,BiLSTM 在处理常含短序列文本的中文电子病历时,如描述患者头晕症状“3天前始有头晕,头昏沉感,并有头痛,睡眠差”,会遇到梯度消失或梯度爆炸的问题,使得模型难以训练和收敛。
解码层根据编码层输出的特征向量序列预测每个位置上的实体标签。然而,在医疗领域存在大量的嵌套命名实体。嵌套命名实体指的是在实体内部还包含其他实体的特殊命名实体,例如,“颅内血管畸形”的实体类型是“疾病”,同时还包含着实体类型为“部位”的“颅内血管”,导致解码器在进行预测时准确度降低。例如,使用 CRF 作为解码器处理嵌套命名实体时会出现难以准确捕捉到实体内部结构的问题。
2 RBBEGP 命名实体识别模型
2.1 嵌入层RoBERTa-wwm-ext
RoBERTa-wwm-ext 是一种基于 BERT 的 Trans⁃former 网络结构的 PLMs。BERT 模型引入了两个任务,任务一是下一语句预测(Next Sentence Prediction,NSP) ,该任务的目标是判断输入 BERT 模型的两个文本段落是否连续。RoBERTa-wwm-ext[13]取消了这一任务,使得模型能够更关注于语言本身的语义关系而非句子结构。任务二是采用掩码语言模型(Mask Lan⁃guage Model, MLM) 的方式随机选择输入序列中的一些词并用特殊符号[MASK]代替,模型根据给定的标签对这些位置应该填入的词进行预测。
RoBERTa-wwm-ext 采用一种基于全词掩码策略(Whole Word Mask,WWM) 的中文训练方式,使模型充分考虑中文字词构成。通过将整个词作为一个单元进行掩码来替代单个字符掩码,模型能够理解中文词汇的含义和全文关系。具体而言,LTP[14]分词工具通过最大熵准则建模标注序列在输入序列情况下的得分函数,使其可以根据输入序列的上下文信息,对词语进行准确的建模和标注。在 RoBERTa-wwm-ext 中采用该分词器将分好的词作为训练目标进行预测,有效解决 BERT 模型只能掩码部分字词的缺点,从而更准确地处理中文文本。
初始文本“高血压可能引起脑梗塞”经分词以及两种掩码策略后的对比如表2所示。对于中文文本,使用 WWM 能够分割到含有完整意义词的位置并保留完整的信息,而 MLM 会丢失词语中的重要信息,对中文 NLP 任务的识别效果产生一定的负面影响。
RoBERTa-wwm-ext 模型结构如图1 所示,其中[CLS]和[SEP]是特殊标记,分别对应句子的开头与结尾,两种标记能够将部分知识编码在权重中,以便分割语句,Ei 是对输入文本进行全词编码后的向量,Ti为对应的输出向量,Trm为Transfomer。
2.2 编码层BiGRU
门控循环单元[15](Gated Recurrent Unit, GRU) 是长短期记忆网络(Long Short-Term Memory, LSTM) 的一种变体,包含一个更新门和一个重置门。更新门确定当前时间步中应保留的信息,重置门确定哪些信息应该被丢弃。这样的门控机制有利于控制信息的流动,以缓解梯度消失或梯度爆炸等问题,更适用于处理短序列数据。
BiGRU由分别用于正向与反向序列建模的两个GRU组成,模型能够结合上下文信息以提高序列数据的建模能力,避免GRU只能捕捉单向时间序列信息的缺陷。第一个GRU单元从序列的第一个时间步开始,向前扫描并生成一个隐藏状态序列;另一个GRU单元从序列的最后一个时间步开始,向后扫描并生成另一个隐藏状态序列。这些隐藏状态序列中蕴含着与输入序列相关的重要信息。各 GRU 单元通过结合更新门与重置门,在每个时间步上有效地更新隐藏状态。
这种机制使得模型能够理解序列数据的上下文信息,捕捉序列中的短期和长期依赖关系,提取关键特征并进行准确的预测或分类。相较于BiLSTM,Bi⁃GRU还减少了参数,模型能够快速收敛。模型结构如图 2 所示。
在i 时刻,隐藏层的输出状态 hi 受到当前时刻的输入向量xi、正向隐藏层输出向量 以及反向隐藏层输出向量的影响。这种双向影响使得模型能够同时考虑到当前时刻文本中的上下文信息,从而更全面地理解序列数据。通过结合正向和反向的隐藏层输出,BiGRU能够更准确地捕捉到序列中的关键模式和依赖关系。
2.3 解码层EGP
指针网络(Pointer Net⁃work) 通常采用两个相互独立的模块来识别实体的头部和尾部,这会导致训练和预测的结果存在较大差异。GP同时考虑了实体的起始和终止位置,利用头部和尾部的特征信息来预测嵌套实体,使得模型能够从全局的角度捕捉文本中的信息。由于 GP的训练和预测过程都是并行的,无须进行递归分母运算且预测时也不需要动态规划,因此更加高效[16]。对于不同类别的实体,GP使用不同的 Head 进行预测,对“颅内血管畸形”嵌套命名实体的预测如图3所示。
定义打分函数sα (i,j ), i 为横坐标,j 为纵坐标。对长度为n 的向量序列h1,h2,...,hn 中的每一个向量生成起始位置表征信息qi,α 和终止位置表征信息kj,α,标记Wq,α 和Wk,α 为变换矩阵,bq,α 和bk,α 为偏置,如式(6)~(7) 所示。
由上式可知,实体类型有多少种就需要有多少个矩阵Wq,α 和Wk,α, Wq,α 和Wk,α ∈ Rd × D,在GP模型中,每增加一种实体类型,则需要新增2Dd个参数。相较于CRF每增加一种实体类型只需要新增加2D个参数而言,这增加了d倍的参数量。常见的做法是D=768,d=64,可见GP模型的参数量远远大于CRF。为此,EGP 引入共享矩阵的理念对参数量进行优化,将命名实体任务识别分解为“抽取”和“分类”两个子任务:1) 在“抽取”阶段认为只有一种实体类型,可以共用一个打分矩阵,即(Wq hi )T (Wk hj )。2)“ 分类”任务中使用“特征拼接+全连接层”来完成,将头尾token的Embedding 相拼接。两项相结合作为新的打分函数,如式(10)所示,此时,每增加一种实体类型所需要的变换矩阵Wα ∈ R2D。
识别中文电子病历中的命名实体过程如下:
1) 输入中文电子病历文本后,首先进行中文分词处理。将能够描述完整含义的字符或词语作为独立部分进行切分,并为语句添加特殊标记。
2) 将分词后的文本转换为词向量,送入RoBERTa-wwm-ext 模型中,以便模型学习句子中的信息,得到结合语义信息的词向量。
3) 将第 2) 步中的输出向量作为BiGRU的输入,通过两种门控机制决定应当被保留或丢弃的信息。
4) 将第3) 步中的向量结合其他特征向量作为输入矩阵输入EGP中进行解码。计算各标签的得分后,选择得分最高的标签序列作为输出,从而得到对应的实体类别。
3 实验结果与分析
3.1 实验参数
实验参数设置如表3所示。
3.2 数据集
本文所采用的数据来源为CCKS2019Yidu-S4K 电子病历数据集,该数据集由专业的医学团队进行人工标注,共有1 000条训练文本与379条测试文本。表4显示了该数据集中包含的疾病与诊断、检查、检验、手术、药物、解剖部位六类实体共计7 179条数据的分布情况。